Mycoplasma laboratorium
Mycoplasma laboratorium es una especie parcialmente sintética, diseñada de bacteria derivados de la genoma de Genitalium del Mycoplasma. Este esfuerzo en biología sintética se realiza en el J. Craig Venter Institute por un equipo de aproximadamente 20 científicos liderados por el Premio Nobel Hamilton Smithy como DNA Investigador Craig Venter y microbiólogo Clyde A. Hutchison III. Genitalium del Mycoplasma fue elegido como lo fue la especie con el menor número de genes conocidos en aquel momento.
En 21 de mayo de 2010, Ciencia informó que el grupo de Venter había sintetizado con éxito el genoma de la bacteria Mycoplasma mycoides de un registro de la computadora y se trasplanta el genoma sintetizado en la célula existente de un Capricolum del Mycoplasma bacteria que había tenido su ADN eliminado. La bacteria "sintética" es viable, es decir, capaces de reproducir miles de millones de veces. (El equipo había planeado originalmente utilizar el M. genitalium bacteria había previamente estado trabajando, pero cambiado a M. mycoides porque la bacteria este último crece mucho más rápido, que se tradujo en experimentos más rápidos.) Científicos que no participaron en la atención de estudio que no es una forma de vida verdaderamente sintética porque su genoma fue puesto en una celda existente.[b 1]
Se estima que el genoma sintético costo US$ 40 millones para hacer y 20 personas más de una década de trabajo.[b 2] A pesar de la controversia, Venter ha atraído más de $ 110 millones en inversiones hasta ahora para Synthetic Genomics, con un futuro acuerdo con Exxon Mobil de $ 300 millones en investigación para el diseño de las algas para combustible diesel.[b 3]
Contenido
- 1 Micoplasma
- 2 Otros géneros
- 3 Proyecto del genoma mínimo
- 4 Trasplante del genoma bacteriano
- 5 Síntesis del cromosoma bacteriano
- 6 Genoma sintético
- 6.1 Marcas de agua
- 7 Preocupaciones y la controversia
- 7.1 Cobertura de prensa
- 7.2 Utilidad
- 7.3 Propiedad intelectual
- 8 JCVI-syn3.0
- 9 Proyectos similares
- 10 Referencias
- 10.1 Fuentes primarias
- 10.2 Prensa popular
- 11 Acoplamientos externos
Micoplasma
Genitalium del Mycoplasma fue elegido como lo fue la especie con el menor número de genes conocidos en aquel momento.
Micoplasma es un género de bacterias de la clase Mollicutes en la división de Tenericutes, caracterizado por la falta de un pared celular (lo que es Gram negativas) debido a su parasitarias o comensales estilo de vida (intracelular y extracelular). En biología molecular, el género ha recibido mucha atención. Además de ser un notorio y difíciles de erradicar (inmune a beta-lactámicos y otros antibióticos) contaminación en mamíferos cultivos celulares,[1] también se ha utilizado como un organismo modelo: la segunda secuencia de genoma bacteriano completo publicado fue el de Genitalium del Mycoplasma, que tiene uno de los más pequeños genomas de los organismos libre-que viven.[2] El M. pneumoniae secuencia del genoma se publicó poco después y se determinó la primera secuencia del genoma de cartilla de caminar de una cósmido Biblioteca en vez de la Escopeta del genoma completo método.[3] Por lo tanto, esta especie fue elegida como modelo para el proyecto de la célula mínima,[4] cataloga el contenido de proteína total de una célula.[5]
Otros géneros
Pelagibacter ubique (un Α-proteobacterium de la orden Rickettsiales) tiene el más pequeño conocido genoma (1.308.759 pares de bases) de cualquier organismo de vida libre y es una de las células más pequeño uno mismo-repliegue conocidas. Es posiblemente la bacteria más numerosas del mundo (tal vez 1028 las células individuales) y, junto con otros miembros de la Grupo SAR11, se estima que constituyen entre un cuarto y mitad de todo bacterianas o arqueales células en el océano.[6] Sin embargo, esta especie fue identificada en 2002 por secuencias de rRNA y fue completamente secuenciado en el año 2005,[7] una extremadamente difícil cultivar especies que no alcanzan una densidad de alto crecimiento,[un 8][9] Además, varias especies recién descubiertas tienen menos genes que M. genitalium, pero muchos genes esenciales que faltan en Hodgkinia cicadicola, Sulcia muelleri, Baumannia cicadellinicola (simbiontes de Cigarras) y Ruddi Ruddii (simbionte del psílido en hackberry peciolo gall, Pachypsylla venusta[10]) pueden ser codificados en el núcleo del anfitrión como estos endosimbiontes están adquiriendo un estatus de organelas en forma similar a las mitocondrias y los cloroplastos.[11]
| nombre de la especie | número de genes | tamaño (Mbp) |
|---|---|---|
| Cicadicola de Candidatus Hodgkinia Dsem [1] | 169 | 0.14 |
| Candidatus Carsonella Carsonella PV [2] | 182 | 0.16 |
| Candidatus Sulcia muelleri GWSS [3] | 227 | 0.25 |
| Candidatus Sulcia muelleri SMDSEM [4] | 242 | 0.28 |
| Buchnera aphidicola Str. Cinara cedri [5] | 357 | 0.4261 |
| Mycoplasma genitalium G37[6] | 475 | 0.58 |
| Candidatus Phytoplasma Mali [7] | 479 | 0.6 |
| Buchnera aphidicola Str. Baizongia pistaciae [8] | 504 | 0.6224 |
| Equitans de Nanoarchaeum Kin4-M [9] | 540 | 0.49 |
Proyecto del genoma mínimo
El equipo empezó con la bacteria M. genitalium, un parásito intracelular obligado cuya genoma consta de 482 genes compuesta por 582.970 pares de bases, dispuesto en la uno circular cromosoma (el más pequeño genoma de cualquier organismo natural conocido que puede ser cultivada en la cultura libre). Luego quitaron sistemáticamente genes para encontrar un conjunto mínimo de 382 genes que pueden sostener la vida.[12] Este esfuerzo también era conocido como el Proyecto del genoma mínimo.
El equipo pretende sintetizar secuencias de la DNA del cromosoma de estos 382 genes. Una vez que ha sido sintetizada una versión del cromosoma 382-gene mínimo, va a ser trasplantado en un M. genitalium célula para crear Mycoplasma laboratorium.
La bacteria de laboratorium resultante de Mycoplasma se espera que sea capaz de replicar sí mismo con su ADN artificial, lo que es el organismo más sintético hasta la fecha, aunque la maquinaria molecular y ambiente químico que permitiera replicar no sería sintético.[b 4]
En diciembre de 2003, el equipo informó un método rápido de sintetizar un genoma desde el principio, producir el genoma de 5386-base de la Bacteriófago Phi X 174 en unas dos semanas.[13] Sin embargo, el genoma de Mycoplasma laboratorium es aproximadamente 50 veces más grande. En enero de 2008, el equipo informó que han sintetizado el cromosoma de base par 582.970 completos de M. genitalium, con pequeñas modificaciones para que no ser infecciosa y puede ser distinguido de la tipo salvaje. Llamaron a este genoma Genitalium del Mycoplasma JCVI-1.0.[14][b 5] El equipo también ha demostrado el proceso de trasplantar un genoma (no sintético) de una Micoplasma especie a otra en junio de 2007.[b 6] En mayo de 2010 demostró que eran capaces de sintetizar el genoma de 1.078.809 pares[15] de Mycoplasma mycoides de rayar y de trasplante en un Capricolum del Mycoplasma célula; el nuevo genoma entonces asumió el control la célula y el organismo nuevo multiplicado.[b 7] El nuevo organismo fue apodado Synthia.
Venter espera finalmente sintetizar bacterias para la fabricación de hidrógeno y biocombustiblesy también para absorber dióxido de carbono y otros gases de efecto invernadero. George M. Church, otro pionero en biología sintética, sostiene que E. coli es un organismo más eficiente que M. genitalium y que la creación de un genoma completamente sintético no es necesario y muy costoso para estas tareas; señala que los genes sintéticos ya han sido incorporados en E. coli para llevar a cabo algunas de las tareas anteriores.[b 5] 28 de junio de 2007, un equipo en el Instituto J. Craig Venter publicó un artículo en Ciencia Express, diciendo que había trasplantado con éxito el ADN natural de una Mycoplasma mycoides bacteria en un Capricolum del Mycoplasma celular, creando una bacteria que se comportó como un M. mycoides.[16]
El 06 de octubre de 2007, Craig Venter anunció en una entrevista con el del Reino Unido El guardián periódico que el mismo equipo había sintetizado una versión modificada de la única cromosoma de Genitalium del Mycoplasma uso de productos químicos. El cromosoma se modificó para eliminar todos los genes que pruebas en bacterias vivas habían demostrado ser innecesarios. El siguiente paso previsto en este proyecto del genoma mínimo es trasplantar el genoma mínimo sintetizado en una célula bacteriana con su antiguo ADN eliminado; la bacteria resultante se llamará Mycoplasma laboratorium. Al día siguiente el canadiense Bioética Grupo, Grupo ETC emitió un comunicado a través de su representante, Pat Mooney, diciendo de Venter "creación" fue "un chasis sobre el que puede crear casi cualquier cosa". El genoma sintetizado aún no había sido trasplantado en una celda de trabajo.[b 4]
En 21 de mayo de 2010, Ciencia informó que el grupo de Venter había sintetizado con éxito el genoma de la bacteria Mycoplasma mycoides de un registro de la computadora y se trasplanta el genoma sintetizado en la célula existente de un Capricolum del Mycoplasma bacteria que había tenido su ADN eliminado. La bacteria "sintética" es viable, es decir, capaces de reproducir miles de millones de veces. El equipo había planeado originalmente utilizar el M. genitalium bacteria había previamente estado trabajando, pero cambiado a M. mycoides porque la bacteria este último crece mucho más rápido, que se tradujo en experimentos más rápidos.[b 2] También han demostrado que el genoma natural de M. mycoides pueden ser transplantados, pero todavía tiene que demostrar que lo mismo podría hacerse M. genitalium.[b 8] Venter lo describe como «la primera especie... que sus padres ser un ordenador».[b 9] La bacteria transformada es apodada"Synthia«por etc. Un portavoz de Venter se ha negado a confirmar cualquier avance en el momento de esta redacción, probablemente porque similares técnicas genéticas como transfección, transformación, transducción de señales y protofection han sido una práctica de investigación estándar durante muchos años.
Ahora que la técnica ha sido probada para trabajar con el M. mycoides genoma, el siguiente proyecto es presumiblemente a la minimización M. genitalium y transplante en una célula para crear el mencionado Mycoplasma laboratorium.
Se anunció la creación de una nueva bacteria sintética en Ciencia en 25 de marzo de 2016. Tiene sólo 473 genes, los genes menores de cualquier libremente vivir organismo. Este rápida crecimiento nueva célula, llamada Syn 3.0, fue creada por trasplantar el genoma de Mycoplasma mycoides, con todo el ADN de los quita en una Mycoplasma capricolum, que había sido vaciado de su propio ADN.
M. genitalium no fue utilizado porque reproduce muy lentamente.
Trasplante del genoma bacteriano
Con el fin de propagar un genoma sintético, la técnica de trasplantar un genoma bacteriano todo intacto en otro tuvo que ser desarrollado. Experimentos pioneros de Oswald Avery en la década de 1940 demostraron que algunas bacterias podrían tomar ADN desnudo,[un 17] y con el advenimiento de técnicas de clonación moleculares Elementos de ADN podrían transformarse en células competentes, por lo general vectores de clonación, unos 5-20 kbp largo e incluso cromosomas artificiales bacterianos se puede mantener. En 2007, el equipo de Venter informó que había logrado transferir el cromosoma de la especie Mycoplasma mycoides Para Capricolum del Mycoplasma por medio de:
- aislar el genoma de M. mycoides: suave lisis de las células atrapadas en agar – agar fundido mezclado con las células y a la izquierda para formar un gel, seguido por gel de electroforesis de pulso del campo y la banda de la talla correcta (circular 1.25Mbp) ser aislado;
- fabricación de las células receptores de M. capricolum competente: crecimiento en medios ricos seguido hambre en medios pobres donde el hambre de nucleótido da lugar a inhibición de la replicación del ADN y el cambio de morfología; y
- glicol de polietileno-mediada por transformación del cromosoma circular a las células de ADN libre seguido por la selección.[16]
El término transformación se utiliza para referirse a la inserción de un vector en una célula bacteriana (por electroporación o heatshock). Aquí, el trasplante se utiliza similares a trasplante nuclear.
El interruptor de M. genitalium Para M. mycoides fue estimulado debido al crecimiento más rápido de esta última.[b 2]
Síntesis del cromosoma bacteriano
Es posible crear secuencias de ADN químicamente ()síntesis de oligonucleótidos) que se obtiene por sucesivas rondas de desprotección y acoplamiento de protegido phosphoramidite nucleótidos geométricamente decrecientes rendimientos a longitud, haciendo inviable secuencias de más de 1kb. Secuencias más largas, la ligadura de la DNA se requiere. En 2008 el grupo de Venter publicó un documento mostrando que había logrado crear un genoma sintético (una copia de M. mycoides secuencia de CP001621) mediante una estrategia jerárquica:[14]
- Síntesis → 1kbp: la secuencia del genoma fue sintetizada por Blue Heron en 1078 1080bp cassettes con 80bp superposición y sitios de restricción NotI (ineficientes pero rara cortador).
- La ligadura → 10kbp: 109 grupos de una serie de 10 cassettes consecutivos eran ligarse y clonados en e. coli en un plásmido y la permutación correcto comprobado por secuenciación, esto seguiría un Distribución geométrica con el número esperado de pruebas de 10.
- Multiplex PCR → 100kbp: 11 grupos de una serie de 10 asambleas de 10kbp consecutivos (crecido en levadura) se unieron por multiplex PCR, usando un par de cartilla para cada conjunto de 10kbp.
- Aislamiento y recombinación → secundario las Asambleas fueron aisladas mediante el método de enchufe arriba y unir y se transforma en Esferoplastos de levaduras sin una secuencia de vectores (presente en Asamblea 900 811).
Genoma sintético
En 2010, utilizando los métodos descritos anteriormente, Venter y sus colegas creó una cepa de Mycoplasma mycoides llamado JCVI-syn1.0 con un genoma sintético.[un 18] Inicialmente la construcción sintética no funcionó, para pin punto el error — que causó un retraso de 3 meses en todo el proyecto[b 2] — se crearon una serie de construcciones semi sintéticas. Dado que el genoma natural trabajado, la causa del crecimiento fue una mutación del mutágeno ' frameshift ' en DnaA, un factor de iniciación de la replicación, que, una vez corregido, trabajó y fue verificada.[un 18]
La construcción de una célula con un genoma sintético fue hecha para probar la metodología, lo que permite más modificados genomas a crearse en el futuro. Para minimizar las fuentes de insuficiencia, el genoma fue creado usando un genoma natural como plantilla. Debido al gran tamaño de un genoma, además de los elementos necesarios para la propagación de levaduras y residuos de los sitios de restricción, varias diferencias están presentes en Mycoplasma mycoides JCVI-syn1.0 en particular una E. coli Transposon IS1 (una infección de la etapa de 10kb) y una duplicación de la 85bp.[un 18]
Sin embargo, el proyecto ha recibido crítica pesada como afirma haber creado un organismo sintético. Esta afirmación surge del hecho de que el genoma sintetizado químicamente en muchos pedazos (método sintético), se unieron por medio de técnicas biológicas moleculares (un método artificial) y trasplantado en el citoplasma de una célula natural (después de algunas generaciones, sin embargo, el contenido de proteína original es indetectable)[citación necesitada]. Las dos especies utilizadas como donante y receptor son del mismo género como las dos especies más distantes, menos las interacciones de la proteína correcta se mantienen, como factores vinculantes y sitios de Unión, que mutan a ()epistasis).[un 19] En consecuencia, Paul Keim (un genetista molecular de Universidad del norte de Arizona en Flagstaff) observa que "hay grandes retos por delante antes de ingenieros genéticos puede mezclar fósforo y diseño completamente el genoma de un organismo desde cero" [b 2] debido a este problema. ADN es la plantilla para la construcción de proteínas y requiere las proteínas como moléculas de ayudante para hacerlo, un enigma del huevo y la gallina resuelto por el Hipótesis del mundo del RNA, por lo tanto sintética DNA desnuda requeriría varias proteínas para crear una célula viable. En 2000 y 2002 equipos sintetizan replicar virus de la hepatitis C (cerca de 9600 nucleótidos de largo)[20] y virus de la poliomielitis (alrededor de 7500 nucleótidos de largo),[un 21] Virus, sin embargo, replican utilizando la maquinaria de expresión de la proteína de host. Además, mientras que el ADN puede ser replicado fácilmente (usando Polimerasa de la DNA), transcripción (usando Polimerasa del RNA) y traducido (con ribosomas y muchos otros factores), todos en vitro, tales reacciones, hasta el momento, utilizan extractos celulares y la mayoría de los componentes no ha sido sintetizados de novo, que es inorgánico o sintético hecho químicos orgánicos solamente.
Marcas de agua

Una característica más publicitada de la Mycoplasma laboratorium es la presencia de secuencias de la marca de agua como una prueba definitiva del logro y como un truco de publicidad, similar a la tradición de arte de chip, inscripciones en porciones no utilizadas de microchips visibles por microscopia electrónica. Las 4 marcas de agua (presente en la figura 1 en material complementario de [un 18]) son mensajes codificados en forma de ADN pares de bases, de 1246, 1081, 1109 y 1222 base pares respectivamente, en natural péptidos los 4 nucleótidos codificar en conjuntos de 3 los 20 aminoácidos naturales por medio de la código genético estándar. Cada aminoácido por la Convención se representa por una letra, pero en la naturaleza no hay nada que Alanina, una molécula, a la letra latina A, una vocal, por lo que este Convenio fue ignorado en las marcas de agua de este último. En el organismo de genoma mínimo la marca de agua se codificaron como aminoácidos, con V como U, tanto en referencia a las inscripciones latinas y la falta de un aminoácido estándar para U que contiene los nombres de los investigadores: (VENTERINSTITVTE, CRAIGVENTER, HAMSMITH, CINDIANDCLYDE, GLASSANDCLYDE). En el organismo sintético, en cambio el alfabeto latino, que en inglés tiene 26 letras, que está cubierto sólo en base 4 con 3 o más dígitos, fue codificada por una codificación no divulgada. La codificación es fijo y 3 dígitos hacen una letra mayúscula o ASCII símbolo, posiblemente asignado al azar (no ASCII tabla, frecuencia o orden de teclado).[b 10] El contenido de las marcas de agua es el siguiente:
- marca de agua 1 un HTML script which reads to a browser as text congratulating the decoder with an email link (mroqstiz@jcvi.org) to click to prove the decoding.
- filigrana 2 contiene una lista de autores y una cita de James Joyce:: "Vivir, errar, caer, triunfar, para recrear la vida de la vida".
- marca de agua 3 contiene más autores y una cita de Robert Oppenheimer (sin acreditar): "Ver las cosas no como son, pero que sean".
- marca de agua 4 contiene aún más autores y una cita de Richard Feynman: "qué no puedo construir, no entiendo".
Preocupaciones y la controversia
Cobertura de prensa
La controversia principal del proyecto es la excesiva cantidad de publicidad que recibió de la prensa debido a la espectacularidad de Venter, en la medida en que Jay Keasling, un pionero biólogo sintético y fundador de Amyris dice "Reglamento único que necesitamos es de boca de mi colega".[b 3]
Utilidad
A pesar de la financiación para aplicaciones prácticas, como por George M. Church, uno de los principales actores en el campo de la biología sintética, algunos cambios se requieren para obtener organismos útiles, tales como producción de biocombustibles o biorremediación.[b 3] Sin embargo, abunda la especulación sobre posibles aplicaciones de un lejano. Venter se es propenso a tales especulaciones, haber solicitado por ejemplo, "¿Qué pasa si podemos hacer algas gusto carne?"[b 3] Si fuera posible crear una célula sintética sin el uso de las células receptores preexistentes, sin embargo, muchas aplicaciones sería alcanzable que sería de otra manera inalcanzable, como una bacteria totalmente reacondicionada que funciona de una manera lógicamente controlada, eliminación de lo que se ha descrito como 'desorden evolutivo'[un 22] — con tasas de mutación, gen categórico arreglo (colinealidad), la posibilidad de añadir nuevos nucleótidos para aumentar la codificación (una hazaña que logró en vitro (PCR)); o con un código genético completamente novedoso, como se ha logrado mediante experimentos en los que unos aminoácidos no-canónico adicionales fueron agregados.
Propiedad intelectual
J. Craig Venter Institute había presentado solicitudes de patente para el genoma de laboratorium del Mycoplasma (el "mínimo genoma bacteriano") en Estados Unidos e internacionalmente en el año 2006.[b 11][b 12][un 23] Esta extensión del dominio de patentes biológicas está siendo cuestionada por la organización del perro guardián Grupo de acción sobre erosión, tecnología y concentración.[b 13]
JCVI-syn3.0
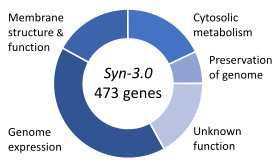
En 2016, el Instituto Venter utiliza genes de JCVI-syn1.0 para sintetizar un genoma más pequeño que llaman JCVI-syn3.0, que contiene 473 genes y 531.560 pares de bases.[2] Originalmente en 1996, después de comparar M. genitalium con otra bacteria pequeña Haemophilus influenzae, Arcady Mushegian y Eugene Koonin propuso que podría ser un conjunto común de 256 genes que podrían ser un conjunto mínimo de genes necesarios para la viabilidad.[3][4] En este nuevo organismo, el número de genes solamente puede ser pared a 473, 149 de los cuales cuyas funciones son totalmente desconocidos.[3] Es el organismo con el más pequeño conjunto de genes conocido Nasuia deltocephalinicola que tiene genes sólo 137,[5] pero el genoma mínimo para cualquier organismo libre-que iba a ser diferente dependiendo de su entorno.[3]
Proyectos similares
De 2002 a 2010, un equipo de la Academia Húngara de Ciencias creó una cepa de Escherichia coli llamado MDS42, que se vende ahora por escarabajo genómica de Madison, WI bajo el nombre de "genoma limpio. E. coli",[b 14] eliminación de donde el 15% del genoma de la cepa parental (e. coli K-12 MG1655) fueron quitado para facilitar la eficiencia de la biología molecular, Elementos es, pseudogenes y fagos, lo que resulta en mejor mantenimiento de genes tóxicos plasmídico, que a menudo son inactivadas por transposones.[24][25][un 26] Maquinaria bioquímica y replicación no fueron alterados.
Referencias
Fuentes primarias
- ^ L joven, Sung J, Stacey G, amos Jr. "detección de Micoplasma en cultivos de células". NAT Protoc. 2010 5(5): 929 – 34. Epub 2010 Abr 22.
- ^ Fraser CM, Gocayne JD, O blanco, et al., (octubre de 1995). "El complemento del gene mínimo del Genitalium del Mycoplasma". Ciencia 270 (5235): 397-403. Bibcode:1995Sci... 270..397F. doi:10.1126/Science.270.5235.397. PMID7569993.
- ^ Himmelreich R, H de Hilbert, H Plagens, Pirkl E, Li BC, Herrmann R (noviembre de 1996). "Completo análisis de la secuencia del genoma de la bacteria Mycoplasma pneumoniae". Investigación de ácidos nucleicos 24 (22): 4420 – 49. doi:10.1093/Nar/24.22.4420. PMC146264. PMID8948633.
- ^ CA de Hutchison, Montague MG (2002). mycoplasmas y el concepto de genoma mínimo. Biología molecular y patogenicidad de los micoplasmas (Razin S, Herrmann R, eds.). Nueva York: Kluwer académico/pleno. págs. 221 – 54. ISBN0-306-47287-2.
- ^ Regula JT, Ueberle B, Boguth G, et al (noviembre de 2000). "hacia un mapa de dos dimensiones proteoma de Mycoplasma pneumoniae". Electroforesis 21 (17): 3765 – 80. doi:10.1002/1522-2683 (200011) 21:17 < 3765::AID-ELPS3765 > 3.0.CO;2-6. PMID11271496.
- ^ Morris RM, et al (2002). "grupo SAR11 domina océano superficie vertical comunidades". Naturaleza 420 (6917): 806-10. Bibcode:2002Natur.420... 806M. doi:10.1038/nature01240. PMID12490947.
- ^ Stephen J. Giovannoni, H. James Tripp, et al (2005). "Racionalización de genoma de una bacteria oceánica cosmopolita". Ciencia 309 (5738): 1242-1245. Bibcode:2005Sci... 309,1242 G. doi:10.1126/Science.1114057. PMID16109880.
- ^ Rappé MS, Connon SA, KL Virgen, Giovannoni SL (2002). "Cultivo de la omnipresente SAR11 vertical Marina clade". Naturaleza 418 (6898): 630 – 33. Bibcode:2002Natur.418... 630R. doi:10.1038/nature00917. PMID12167859.
- ^ HJ Tripp Kitner JB, MS de Schwalbach, Dacey JW, Wilhelm LJ, Giovannoni SJ (10 de abril de 2008). "bacterias marinas SAR11 requieren azufre reducido exógeno para el crecimiento". Naturaleza 452 (7188): 741 – 4. Bibcode:2008Natur.452... 741T. doi:10.1038/nature06776. PMID18337719.
- ^ Nakabachi, A.; Yamashita, A.; Toh, H.; Ishikawa, H.; Dunbar, H. E.; Moran, A. N.; Hattori, M. (2006). "El genoma del Kilobase 160 de la bacteriano Endosymbiont Ruddii". Ciencia 314 (5797): 267. doi:10.1126/Science.1134196. PMID17038615.
- ^ McCutcheon, J. P.; McDonald, R. B.; Morán, N. A. (2009). "Evolución convergente de roles metabólicos en bacterias simbiontes Co de insectos". Actas de la Academia Nacional de Ciencias 106 (36): 15394-15399. Bibcode:2009PNAS... 10615394M. doi:10.1073/pnas.0906424106. PMC2741262. PMID19706397.
- ^ Vidrio, John I.; Nacyra Assad-García; Nina de Alperovich; Ahora Yooseph; Matthew R. Lewis; Mahir Maruf; Clyde A. Hutchison; Hamilton O. Smith; J. Craig Venter (2006-01-10). "Genes esenciales de una bacteria mínima". Actas de la Academia Nacional de Ciencias 103 (2): 425 – 430. Bibcode:2006PNAS... 103..425G. doi:10.1073/pnas.0510013103. PMC1324956. PMID16407165. 2007-10-16.
- ^ Smith, Hamilton O.; Clyde A. Hutchison; Cynthia Pfannkoch; J. Craig Venter (2003-12-23). "generación de un genoma sintético por Asamblea de todo el genoma: bacteriófago {phi} X174 de oligonucleótidos sintéticos". Actas de la Academia Nacional de Ciencias 100 (26): 15440-15445. Bibcode:2003PNAS... 10015440S. doi:10.1073/pnas.2237126100. PMC307586. PMID14657399. 2007-10-08.
- ^ a b Gibson, B; Clyde A. Hutchison; Cynthia Pfannkoch; J. Craig Venter; et al (2008-01-24). "Completar la síntesis química, montaje y reproducción de un genoma del genitalium del Mycoplasma". Ciencia 319 (5867): 1215-20. Bibcode:2008Sci... 319,1215 G. doi:10.1126/Science.1151721. PMID18218864. 2008-01-24.
- ^ "Completar el genoma de sintético micoplasma mycoides JCVI-syn1.0".
- ^ a b C de Lartigue, vidrio JI, Alperovich N, Pieper R, Parmar PP, Hutchison CA 3, Smith HO, Venter JC (03 de agosto de 2007). "trasplante del genoma en bacterias: cambiar una especie a otra". Ciencia 317 (5838): 632-8. Bibcode:2007Sci... 317..632 L. doi:10.1126/Science.1144622. PMID17600181.
- ^ Avery OT, Macleod CM, McCarty M (01 de febrero de 1944). "Estudios sobre la naturaleza química de la sustancia que induce la transformación de los tipos de neumococos: inducción de la transformación por una fracción de ácidos Desoxyribonucleic aislada del tipo III del neumococo". J Exp Med. 79 (2): 137-58. doi:10.1084/Jem.79.2.137. PMC2135445. PMID19871359.
- ^ a b c d Gibson, G. D.; Glass, J. I.; Lartigue, C.; Noskov, V. N.; Chuang, R. Y.; Algire, M. A.; Dobladoras, g. A.; Montague, M. G.; MA, L.; Moodie, M. m..; Merryman, C.; Vashee, S.; Krishnakumar, R.; Assad-García, N.; Andrews-Pfannkoch, C.; Denisova, e. A.; Young, L.; Qi, Z. Q.; Segall-Shapiro, T. H.; Calvey, C. H.; Parmar, P. P.; Hutchison, C. A.; Smith, H. O.; Venter, J. C. (20 de mayo de 2010). "La creación de una célula bacteriana controlada por un genoma químicamente sintetizado". Ciencia 329 (5987): 52 – 56. Bibcode:2010Sci... 329... 52G. doi:10.1126/Science.1190719. PMID20488990.
- ^ Povolotskaya, es; Kondrashov, FA (Jun de 2010). espacio y la continua expansión del universo de proteína. Naturaleza 465 (7300): 922 – 6. doi:10.1038/nature09105.
- ^ Destrozo de la KJ, Kolykhalov AA, arroz CM (08 de diciembre de 2000). "Eficiente iniciación de la replicación del ARN del VHC en cultivos celulares". Ciencia 290 (5498): 1972 – 4. Bibcode:2000Sci... 290.1972B. doi:10.1126/Science.290.5498.1972. PMID11110665.
- ^ Violonchelo, J; Pablo, AV; Wimmer, E (agosto de 2002). "síntesis química del cDNA del poliovirus: generación de virus infeccioso en ausencia de la plantilla natural". Ciencia 297 (5583): 1016-8. doi:10.1126/Science.1072266. PMID12114528.
- ^ Tawfik, Dan S (2010). Biología desordenado y el origen de innovaciones evolutivas. Nature Chemical Biology 6: 692-696. doi:10.1038/nchembio.441.
- ^ Estados Unidos solicitud de patente: 20070122826
- ^ K Umenhoffer, Fehér T, Balikó G, Ayaydin F, Pósfai J, Blattner FR, Pósfai G (2010). "A menor evolución de Escherichia coli MDS42, un chasis celular es para aplicaciones de biología molecular y sintético". Fábricas de la célula de Microb 9: 38. doi:10.1186/1475-2859-9-38. PMC2891674. PMID20492662.
- ^ Pósfai G, Plunkett G 3, Fehér T, Frisch D, GM Keil, Umenhoffer K, Kolisnychenko V, Stahl B, Sharma SS, de Arruda M, V Burland, Harcum SW, Blattner FR (2006). "Propiedades emergentes de genoma reducido Escherichia coli". Ciencia 312 (5776): 1044-6. Bibcode:2006Sci... P 312,1044. doi:10.1126/Science.1126439. PMID16645050.
- ^ Kolisnychenko V, Plunkett G 3, arenque CD, T Fehér, Pósfai J, Blattner FR, Pósfai G (abril de 2002). "Ingeniería de un genoma reducido de coli de Escherichia". Res de genoma 12 (4): 640-7. doi:10.1101/gr.217202. PMC187512. PMID11932248.
Prensa popular
- ^ «científicos crean sintéticos de la célula, versión 1.0".
- ^ a b c d e E Pennisi (mayo de 2010). "genómica. Genoma sintético trae nueva vida a la bacteria" (PDF). Ciencia 328 (5981): 958-9. doi:10.1126/Science.328.5981.958. PMID20488994.
- ^ a b c d Andrew Pollack, su estrategia corporativa: El método científico, NYTimes, 04 de septiembre de 2010
- ^ a b Pilkington, Ed (06 de octubre de 2009). «estoy creando vida artificial, declara pionero de gen de Estados Unidos». Londres: El guardián. Nov 23 2012.
- ^ a b Pieza más larga de ADN sintético todavía, Noticias americanas científicas, 24 de enero de 2008
- ^ Bamboleo, Nicholas (2007-06-29). "Científicos trasplantar el genoma de bacterias". El New York Times. ISSN0362-4331. 2007-12-28.
- ^ Henderson, Mark (21 de mayo de 2010). «Científicos crean vida artificial en laboratorio». Los tiempos de (Londres).
- ^ Roberta Kwok (2010). "genómica: artesanos del ADN". Naturaleza 468 (7320): 22 – 5. Bibcode:2010Natur.468... 22K. doi:10.1038/468022a. PMID21048740.
- ^ "cómo los científicos hacen ' vida artificial'". Noticias de BBC. 2010-05-20. 2010-05-21.
- ^ Ken Shirriff (2010-06-10). "Usando el arco para descifrar el secreto de Venter filigrana de ADN". Blog de Ken Shirriff. 2010-10-29.
- ^ "Vida artificial: patente pendiente", El economista, 14 de junio de 2007. Obtenido 07 de octubre de 2007.
- ^ Roger Highfield "Microbio artificial ' para crear biocombustibles sin fin'", Telégrafo, 08 de junio de 2007. Obtenido 07 de octubre de 2007.
- ^ "Primera patente reclamado en la forma de vida artificial y desafió", Ciencia del mundo, 07 de junio de 2007. Obtenido 07 de octubre de 2007.
- ^ "escarabajo genómica LLC. Sitio de web de la compañía.".
- ^ Hutchison, Clyde a.; Chuang, Ray-Yuan; Noskov, Vladimir N.; Assad-Garcia, Nacyra; Deerinck, Thomas J.; Ellisman, Mark H.; Gill, John; Kannan, Krishna; Karas, J. Bogumil (2016-03-25). "Diseño y síntesis de un genoma bacteriano mínimo". Ciencia 351 (6280): aad6253. doi:10.1126/Science.aad6253. ISSN0036-8075. PMID27013737.
- ^ Primera célula bacteriana sintética mínima. Web de Astrobiología. 24 de marzo de 2016.
- ^ a b c Yong, Ed (24 de marzo de 2016). "La cosa misteriosa acerca de una maravillosa nueva célula sintética".
- ^ R. de Arcady Mushegian y Eugene V. Koonin (septiembre de 1996). "Un gene mínimo fijado para la vida celular derivada de la comparación de genomas bacterianos completos" (PDF). E.e.u.u. el proc. nacional Acad. SCI. 93: 10268 – 10273. PMID8816789.
- ^ Zimmer, Carl. "Y los genomas manten...". National Geographic.
Acoplamientos externos
- J. Craig Venter Institute: Grupos de investigación
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||