FRÍO-PCR
FRÍO-PCR (co-amplificación en lower dtemperatura de enaturation-POLIMERIZACIÓN EN CADENA) es la Reacción en cadena de polimerasa Protocolo (PCR) que enriquece la variante alelos de una mezcla de tipo salvaje y mutación de-que contiene DNA. La capacidad preferentemente amplificar e identificar los alelos de la minoría y bajo nivel somática Mutaciones de la DNA en presencia de alelos de tipo salvaje exceso es útil para la detección de mutaciones. Detección de mutaciones es importante en el caso de temprano cáncer detección de tejido biopsias de y fluidos corporales tales como plasma de sangre o suero, evaluación de residuos enfermedad después de la cirugía o quimioterapia, estadificación de la enfermedad y perfiles moleculares de pronóstico o adaptando la terapia a cada paciente y seguimiento del resultado de la terapia y cáncer remisión o recaída. PCR común amplificará la major (wildtype) y menores (mutantes) alelos con la misma eficiencia, ocluyendo la capacidad de detectar fácilmente la presencia de mutaciones de bajo nivel. La capacidad para detectar una mutación en una mezcla de variante/wildtype ADN es valiosa porque esta mezcla de variante que DNAs pueden ocurrir cuando con una muestra heterogénea – como a menudo es el caso de biopsias de cáncer. Actualmente, se utiliza PCR tradicional junto con un número de diferentes ensayos aguas abajo para genotyping o la detección de somática mutaciones. Estos pueden incluir el uso de ADN amplificado para RFLP Análisis, MALDI-TOF genotipificación (matriz asistida por láser-desorción – tiempo de vuelo) o secuencia directa para la detección de mutaciones por Sanger secuenciación o Pirosecuenciación. Sustitución de PCR tradicional con frío-PCR para estos ensayos abajo aumentará la fiabilidad en la detección de mutaciones de muestras mixtas, incluyendo tumores y fluidos corporales.
Contenido
- 1 Resumen del método de frío-PCR
- 1.1 Lleno de frío-PCR
- 1.2 FRÍO rápido-PCR
- 2 Uso de frío-PCR hasta la fecha
- 2.1 RFLP y PCR de frío
- 2.2 Secuenciación de Sanger y frío-PCR
- 2.3 Pirosecuenciación y frío-PCR
- 2.4 MALDI-TOF y frío-PCR
- 2.5 QPCR y frío-PCR
- 3 Ventajas de COLD-PCR
- 4 Desventajas de frío-PCR
- 5 Historia
- 6 Alternativas
- 7 Véase también
- 8 Referencias
Resumen del método de frío-PCR

El principio subyacente de frío-PCR es que solo nucleótido desajustes alterarán ligeramente la temperatura de fusión (Tm) de la DNA de doble hebra. Dependiendo del contexto de la secuencia y posición de la unión mal hecha, Tm cambia de 0.2-1.5 ° C (0.36 2.7 ° F). son comunes para las secuencias hasta 200bp o superior. Sabiendo esto los autores del protocolo tomaron ventaja de dos observaciones:
- Cada ADN de doble hebra tiene una "temperatura crítica' (Tc) inferior a su Tm. La eficiencia de amplificación de PCR mensurable cae por debajo de la Tc.
- La Tc es dependiente de la secuencia de ADN. Dos fragmentos de la DNA de la plantilla difieren por sólo uno o dos nucleótidos desajustes tendrán eficiencias de amplificación diferentes si el paso de la desnaturalización de PCR se establece en el Tc.
Teniendo estos principios en mente los autores desarrollaron el siguiente protocolo general:
- Etapa de desnaturalización. ADN está desnaturalizado a alta temperatura, generalmente a 94 ° C (201 ° F).
- Etapa de recocido intermedio. Establecer una temperatura de recocido intermedia que permite la hibridación del alelo mutante y wildtype DNA uno al otro. Porque el alelo mutado DNA forma la minoría de la DNA en la mezcla sean más propensos a formar desajuste del heterodúplex ADN con el ADN de tipo salvaje.
- Etapa de fusión. Estos heterodúplex se funde más fácilmente a temperaturas más bajas. Por lo tanto son selectivamente desnaturalizados en el Tc.
- Etapa de recocido de la cartilla. El ADN del homo-duplex preferentemente permanecerá doble trenzado y no esté disponible para el recocido de la cartilla.
- Etapa de extensión. Polimerasa de la DNA se extenderá a la plantilla de ADN complementario. Desde del heterodúplex DNA se utiliza como plantilla, una mayor proporción de la variante menor ADN se amplificarán y estar disponible para posteriores rondas de PCR.
Hay dos formas de frío-PCR que se han desarrollado hasta la fecha. Full frío-PCR y PCR de frío rápido.
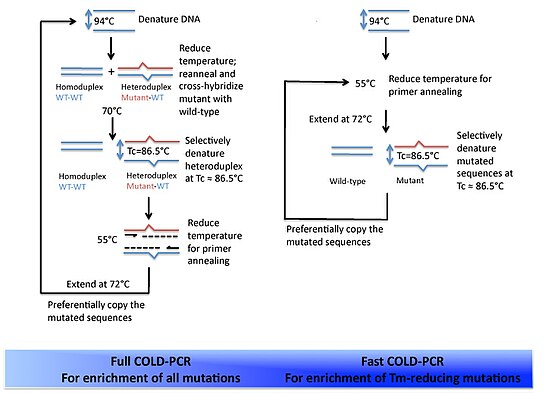
Lleno de frío-PCR
Completo frío-PCR es idéntico al protocolo descrito anteriormente. Estas cinco etapas se utilizan para cada ronda de amplificación.
FRÍO rápido-PCR
FRÍO rápido-PCR diferencia de completo frío-polimerización en cadena en que se omiten las etapas de recocido intermedias y desnaturalización. Esto es porque, en algunos casos, la amplificación preferencial de la mutante de ADN es tan grande que asegura la formación de la mutante/wildtype del heterodúplex ADN no es necesario. Así puede ocurrir la desnaturalización en el Tc, proceda a recocido de la cartilla y extensión entonces mediada por la polimerasa. Cada ronda de amplificación incluye estas tres etapas en ese orden. Mediante la utilización de la más baja temperatura de desnaturalización, la reacción se discrimina hacia los productos con la Tm inferior – es decir, los alelos variantes. FRÍO rápido-PCR produce resultados mucho más rápidos debido al protocolo acortado. Sin embargo, es importante tener en cuenta que frío completo-PCR es esencial para la amplificación de todas las mutaciones posibles en la mezcla inicial de ADN.
Doble ronda fría-PCR es una versión modificada de la rápida frío-PCR. Durante la segunda ronda de la rápida frío-PCR se utilizan cebadores anidados. Esto mejora la sensibilidad de detección de mutación en comparación a una sola ronda rápida frío-PCR.[1]
Uso de frío-PCR hasta la fecha
FRÍO-PCR se ha utilizado para mejorar la fiabilidad de un número de diferentes ensayos que tradicionalmente utilizan PCR convencional.

RFLP y PCR de frío
Un polimorfismo de longitud de fragmentos de restricción resulta en el escote (o falta) de la DNA para una mutación específica por una enzima de restricción seleccionada que se desdoblan el DNA de tipo salvaje. En un estudio usando una mezcla de tipo salvaje y mutación que contiene ADN amplificado por PCR o frío-PCR, frío-PCR anterior análisis RFLP fue demostrado para mejorar la detección de mutación 10-20 veces.[2]
Secuenciación de Sanger y frío-PCR
Sanger secuenciación recientemente se utilizó para evaluar el enriquecimiento del mutante de ADN de una mezcla de 1:20 mutante: wildtype ADN. La variante que contiene una mutación del ADN se obtuvo de una cáncer de mama línea celular sabido que contienen p53 mutaciones.[1] Comparación de cromatogramas de secuenciación de Sanger indicó que el alelo mutante doble 13 enriquecido cuando frío-PCR se utilizó en comparación a la PCR tradicional solamente.[1] Esto fue determinado por el tamaño de los picos en el cromatograma en la ubicación del alelo variante.
Así, frío-PCR fue utilizada para detectar las mutaciones de p53 de pulmón-adenocarcinoma muestras. El estudio fue capaz de detectar bajo nivel 8 (bajo 20% abundancia) mutaciones que probablemente habría perdidas usando métodos convencionales que no enriquecen para variante de la secuencia de ADN.
Pirosecuenciación y frío-PCR
Similar a su uso en directo Sanger secuenciación con pirosecuenciación frío-PCR ha demostrado ser capaz de detectar mutaciones que tenían una prevalencia de 0.5-1% de las muestras utilizado.[3] FRÍO-PCR fue utilizada para detectar mutaciones de KRAS y p53 por pirosecuenciación y fue demostrado para superar a la PCR convencional en ambos casos.
MALDI-TOF y frío-PCR
El mismo grupo de investigación que desarrolló frío-PCR y utiliza para comparar la sensibilidad de la PCR regular para genotipificación secuenciación, RFLP y pirosecuenciación, directo de Sanger también funcionó un estudio similar usando MALDI-TOF como una aplicación de abajo para la detección de mutaciones. Sus resultados indicaron que frío-PCR podría enriquecer secuencias de mutación de una mezcla de ADN de 10-100 veces y que las mutaciones con una prevalencia inicial de 0.1-0.5% sería detectable.[4] Comparado con la tasa de detección de bajo nivel de 5-10% con PCR tradicional.
QPCR y frío-PCR
FRÍO-PCR ejecutar en un PCR cuantitativa de la máquina, usando TaqMan sondas específicas para una mutación, fue demostrado para aumentar la diferencia medida entre muestras de mutante y de tipo salvaje.[4]
Ventajas de COLD-PCR
- Solo paso método capaz de enriquecer ambos alelos conocidos y desconocidos minoría independientemente del tipo de mutación y posición.
- No requiere reactivos adicionales ni maquinaria especializada. Por lo tanto no se incrementa el costo.
- Mejor que PCR convencional para la detección de mutaciones en un mezclado de la muestra.
- No aumentó significativamente experimento duración comparado con PCR convencional.
Desventajas de frío-PCR
- Tc óptima debe ser medido y determinado para cada amplicón. Agregar un paso adicional a los procedimientos convencionales basados en PCR.
- Requisito para el control de la temperatura de desnaturalización precisas durante PCR a ± 0.3 ° C (° F 0,54).
- Una temperatura crítica adecuada puede no estar disponible que distingue entre secuencias de ADN mutante y wildtype.
- Restringida al análisis de secuencias más pequeñas de aproximadamente 200bp.
- Vulnerables a los errores introducidos por la polimerasa.
- Variable total mutación enriquecimiento que depende de la posición y nucleótidos sustitución de ADN.
- No hay garantía de que todas las mutaciones bajo nivel serán enriquecidas preferencialmente.
Historia
FRÍO-PCR fue descrito originalmente por Li et al.. en un artículo de Nature Medicine publicado en 2008 del grupo de laboratorio de Dr. Mike Makrigiorgos en el Dana Farber Cancer Institute de Harvard Medical School.[2] Como resumido anteriormente, la tecnología se ha utilizado en un número de experimentos de la prueba de principio y experimentos diagnóstico de la investigación médica.
Recientemente, la tecnología de frío-PCR ha sido licenciada por Transgenomic, Inc. Los términos de licencia incluyen los derechos exclusivos para comercializar la tecnología combinada con Sanger secuenciación. Los planes son desarrollar aplicaciones comerciales que permitan la detección rápida de alta sensibilidad de bajo nivel somáticas y mitocondriales de mutaciones de ADN.[5]
Alternativas
Otras tecnologías están disponibles para la detección de mutaciones de la DNA de minorías, y estos métodos pueden ser segregados en su capacidad para enriquecer para detectar mutaciones ya sea conocidas o desconocidas.[6]

Véase también
- POLIMERIZACIÓN EN CADENA
- Genotyping
- Polimorfismo de nucleótido
- Genotipado SNP
Referencias
- ^ a b c Li J, Milbury CA, Li C, Makrigiorgos GM (noviembre de 2009). "Doble ronda fría-polimerización en cadena-basado Sanger secuenciación identifica un nuevo espectro de mutaciones de bajo nivel en adenocarcinoma de pulmón". Mutación humana 30 (11): 1583 – 90. doi:10.1002/humu.21112. PMC2784016. PMID19760750.
- ^ a b Li J, Wang L, H Mamon, Kulke MH, Berbeco R, Makrigiorgos GM (mayo de 2008). Reemplazando la PCR con frío-PCR enriquece variantes secuencias de ADN y redefine la sensibilidad de las pruebas genéticas. Medicina de la naturaleza 14 (5): 579-84. doi:10.1038/nm1708. PMID18408729.
- ^ Zuo Z, Chen SS, Chandra PK et al (agosto de 2009). "Aplicación de frío-PCR para mejor detección de mutaciones de KRAS en muestras clínicas". Patología moderna 22 (8): 1023 – 31. doi:10.1038/modpathol.2009.59. PMID19430420.
- ^ a b Li J, Makrigiorgos GM (abril de 2009). "Frío-PCR: una nueva plataforma para la detección de la mutación altamente mejorada en el cáncer y las pruebas genéticas". Transacciones de la sociedad bioquímicos 37 (2): 427-32. doi:10.1042/BST0370427. PMID19290875.
- ^ Equipo editorial de Laboratorytalk (Oct de 2009). «Transgenomics licencias de tecnología fría-PCR».
- ^ Milbury CA, Li J, Makrigiorgos GM (abril de 2009). "Métodos basados en PCR para el enriquecimiento de la minoría alelos y mutaciones". Química clínica 55 (4): 632 – 40. doi:10.1373/clinchem.2008.113035. PMC2811432. PMID19201784.