Dímeros de primer
A Dímero de la cartilla (PD) es un subproducto potencial en POLIMERIZACIÓN EN CADENA, un método biotecnológico común. Como su nombre lo indica, consiste en un PD cartilla de moléculas que se han unido)cruzado por hibridación) entre sí por cadenas de complementarias bases en las cartillas. Como resultado, la Polimerasa de la DNA amplifica la PD, llevando a la competencia de los reactivos PCR, así potencialmente inhibir la amplificación de la DNA destinados a la amplificación por PCR de secuencia. En PCR cuantitativa, PDs pueden interferir con la cuantificación exacta.
Contenido
- 1 Mecanismo de formación
- 2 Detección de
- 3 Prevenir la formación de dímeros de primer
- 3.1 Software de diseño de la cartilla
- 3.2 PCR Hot-start
- 3.3 Modificaciones estructurales de los primers
- 3.4 Prevención de la adquisición de la señal de dímeros de primer
- 4 Referencias
- 5 Enlaces externos
Mecanismo de formación
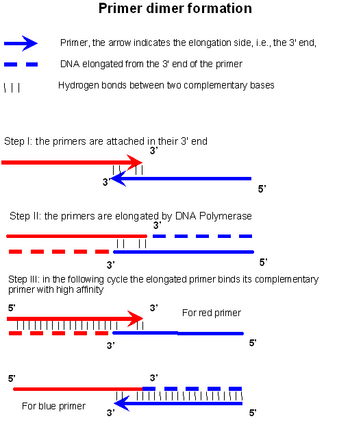
Dímero de la cartilla es formado y amplificado en tres pasos. En el primer paso, dos cebadores hibridan en sus extremos respectivos de 3' (paso en la figura). Si esta construcción es estable, la Polimerasa de la DNA se unen y extiende los cebadores según la secuencia complementaria (paso de II en la figura). Un factor importante que contribuye a la estabilidad de la construcción en el paso es una alta Contenido de GC en el extremos 3' y la longitud de la superposición. El tercer paso se produce en el próximo ciclo, cuando se utiliza un solo filamento del producto de paso II como una plantilla que fresco cartillas recuecen conduce a la síntesis de más producto de PD.[citación necesitada]
Detección de
Dímeros de la cartilla pueden ser visibles después de electroforesis en gel de el producto de PCR. PDs en bromuro de etidio-geles teñidos se consideran típicamente como una banda de par bajo (bp) o frotis de moderada a alta intensidad de 30-50 y distinguible de la banda de la secuencia de destino, que es típicamente más de 50 bp.
En PCR cuantitativa, PDs pueden detectarse por Análisis de la curva de fusión con intercalando tales como tintes, SYBR Green I, un tinte no específico para la detección de ADN de doble hebra. Porque generalmente consisten en secuencias cortas, el PDs desnaturalícelo a menor temperatura que la secuencia de destino y por lo tanto se puede diferenciar por sus características de la curva de fusión.
Prevenir la formación de dímeros de primer
Un enfoque para prevenir la PDs consta de optimización fisico-químicos del sistema PCR, es decir, cambiando las concentraciones de primers, cloruro de magnesio, nucleótidos, fuerza iónica y la temperatura de la reacción. Este método es algo limitada por las características físico-químicas que también determinan la eficiencia de amplificación de la secuencia target en la PCR. Por lo tanto, reducir la formación de PDs también puede resultar en reducida eficiencia de la PCR. Para superar esta limitación, otros métodos pretenden reducir la formación de PDs, incluyendo el primer diseño y el uso de diferentes sistemas de PCR enzimas o reactivos.[citación necesitada]
Software de diseño de la cartilla
Cartilla-diseño de software utiliza algoritmos que compruebe el potencial de la DNA estructura secundaria formación y recocido de los primers a sí mismo o dentro de pares de la cartilla. Parámetros físicos que son tomados en cuenta por el software son uno mismo-complementariedad potencial y contenido de GC de los iniciadores; temperatura de fusión similar de los iniciadores; y ausencia de secundaria las estructuras, tales como vástago-lazos, en el ADN del secuencia de destino.[1]
PCR Hot-start
Porque los iniciadores están diseñados para tener baja complementariedad entre sí, pueden templar (paso en la figura) sólo a baja temperatura, por ejemplo, la temperatura ambiente, tales como durante la preparación de la mezcla de reacción. Aunque polimerasas de la DNA usados en PCR son más activos alrededor de 70° C, tienen alguna actividad de polimerización también en temperaturas más bajas, que pueden causar la síntesis de ADN primers después de recocer uno al otro.[citación necesitada] Varios métodos se han desarrollado para prevenir la formación de PDs hasta que la reacción alcanza la temperatura de trabajo (60-70° C), y estos incluyen inhibición inicial de la polimerasa de la DNA, o separación física de la reacción de los componentes de la reacción hasta que la mezcla de reacción alcance las temperaturas más altas. Estos métodos se conocen como caliente-empiece PCR.
Cera:: en este método la enzima espacial se separa de la mezcla de reacción por la cera que se derrite cuando la reacción alcanza altas temperaturas.[2]
Liberación lenta de magnesio:: ADN polimerasa requiere iones de magnesio para la actividad,[3] así que el magnesio químicamente se separa de la reacción mediante la Unión a un compuesto químico y se libera en la solución sólo en alta temperatura [4]
Unión no covalente del inhibidor de la:: en este método una péptido, anticuerpo[5] o aptámeros[6] son forma no covalente enlazado a la enzima a bajas temperaturas e inhiben su actividad. Después de una incubación de 1 a 5 minutos a 95° C, el inhibidor se libera y comienza la reacción.
Polimerasa de Taq sensibles al frío:: es una ADN polimerasa modificada casi sin actividad a baja temperatura.[7]
Modificación química:: en este método es una pequeña molécula covalentemente a la cadena lateral de una Del aminoácido En sitio activo de la polimerasa de la DNA. La pequeña molécula es liberada de la enzima por la incubación de la mezcla de reacción durante 10 – 15 minutos a 95° C. Una vez que se libera la molécula pequeña, la enzima se activa.[8]
Modificaciones estructurales de los primers
Otro enfoque para prevenir o reducir la formación de PD es modificando los iniciadores para que recocido con ellos mismos o entre sí no causa la extensión.
MANOS (HOmo-Tag Assisted NtintaDImer System[9]): se añade una cola de nucleótidos complementaria al extremo 3' del primer al extremo 5' de la cartilla. Debido a la proximidad de la cola 5' recuece en el extremo 3' de la cartilla. El resultado es un lazo del vástago cartilla que excluye recocido que implican menor superposición, pero permite el recocido de la cartilla a su secuencia complementaria totalmente en blanco.
Cartillas de quiméricos:: algunas bases del ADN en la cartilla se reemplazan con ARN bases, creando un secuencia de quimérico. La temperatura de fusión de una quimérica secuencia con otra secuencia quimérica es inferior a la de secuencia ADN quimérico. Esta diferencia permite ajustar la temperatura de recocido que se recueza la cartilla a su secuencia de destino, pero no para otras imprimaciones quiméricas.[10]
Prevención de la adquisición de la señal de dímeros de primer
Mientras que los métodos anteriores están diseñados para reducir la formación de PD, otro enfoque intenta minimizar la señal generada en PDs en PCR cuantitativa. Este enfoque es útil como hay pocos PDs formadas y su efecto inhibitorio sobre la acumulación de producto es menor.
Cuatro pasos PCR:: utilizado cuando se trabaja con colorantes no específicos, como el SYBR Green I. Se basa en la diferente longitud y por lo tanto, diferente temperatura de fusión de las PDs y la secuencia de destino. En este método la señal es adquirida por debajo de la temperatura de fusión de la secuencia de destino, pero por encima de la temperatura de fusión de las PDs.[11]
Sondas específicas de secuencia: TaqMan y Marcador molecular sondas de generan la señal sólo en presencia de su secuencia (complementarias) de destino, y esta mayor especificidad impide la adquisición de señales (pero no es posibles efectos inhibitorios en la acumulación de producto) del PDs.
Referencias
- ^ La página de diseño de la cartilla del Leiden University Medical Center
- ^ Chou, Quin; Russell, Marion; Abedul, David E.; Raymond, Jonathan; Bloch, voluntad (1992). "Prevención de la dimerización de mal cebado y cartilla de pre-PCR mejora amplificaciones de bajo número de copias". Investigación de ácidos nucleicos 20 (7): 1717-23. doi:10.1093/Nar/20.7.1717. PMC312262. PMID1579465.
- ^ Yang, ahora; Arora, Karunesh; Beard, William A.; Wilson, Samuel H.; Schlick, Tamar (2004). "Papel crítico de iones de magnesio en la Asamblea de cierre y sitio activo de la polimerasa de la DNA beta". Revista de la sociedad química americana 126 (27): 8441-53. doi:10.1021/ja049412o. PMID15238001.
- ^ Solicitud de patente de EEUU número 2007/0254327
- ^ Número de patente US 5338671
- ^ Número de patente US 6183967
- ^ Número de patente US 6214557
- ^ Número de patente US 5677152
- ^ Brownie, Jannine; Shawcross, Susan; Theaker, Jane; Whitcombe, David; Ferrie, Richard; Newton, Clive; Poco, Stephen (1997). "La eliminación de la acumulación de dimero cartilla en PCR". Investigación de ácidos nucleicos 25 (16): 3235 – 41. doi:10.1093/Nar/25.16.3235. PMC146890. PMID9241236.
- ^ «Quiméricos iniciadores para reacciones de amplificación de ácidos nucleicos mejorada». Lente de patente.
- ^ Cuatro pasos PCR
Enlaces externos
«Software en línea para la predicción de dímero de primer». 3.1 OligoAnalyzer. Tecnologías del ADN integrado.
"Diseño de la cartilla. ¿Qué es la cartilla-dimer?. Video de YouTube.