Oligonucleótidos específicos de alelo
Un oligonucleótidos específicos de alelo (ASO) es una pieza breve de sintético DNA complementaria a la secuencia de una ADN diana variable. Actúa como un punta de prueba la presencia del destino en una Mancha blanca /negra meridional ensayo o, más comúnmente, en el más simple Blot dot ensayo. Es una herramienta común utilizada en las pruebas genéticas, medicina forense, y Biología molecular investigación.
Un ASO es típicamente una oligonucleótidos de 15 a 21 bases nucleótidas en longitud. Se diseña (y utilizado) de una manera que hace que sea específico para una única versión, o alelo, de la DNA probada. La longitud de la ASO, que lo cadena es y el las condiciones por las cuales es obligado a (y lavado de) el ADN todos Diana desempeñan un papel en su especificidad. Estas puntas de prueba generalmente pueden ser diseñados para detectar una diferencia de tan poco como 1 base en la secuencia genética del objetivo, una capacidad básica en el análisis de polimorfismos de nucleótido (SNPs), importante en Análisis del genotipo y de la Proyecto genoma humano. Para ser detectado después de que ha limitado a su destino, la ASO debe etiquetarse con una etiqueta radiactiva, enzimática o fluorescente. El Análisis de metilación de Illumina la tecnología aprovecha de ASO para detectar una diferencia de pares (citosina y timina) para medir la metilación en un sitio específico de la GPC.
Contenido
- 1 Ejemplo
- 2 Alternativas
- 3 Historia
- 4 Referencias
- 5 Enlaces externos
Ejemplo

La enfermedad humana anemia de células falciformes es causada por una genética mutación de En codón para la sexta del aminoácido de la proteína de la sangre beta de la hemoglobina. Los códigos de secuencia G-A-G ADN normales para el aminoácido glutamato, mientras que la mutación cambia el medio adenina a un timina, llevando a la secuencia de G-T-G (G-U-G en la mRNA). Esto altera secuencia sustitutos un valina en la proteína final, distorsionando su estructura.
Para comprobar la presencia de la mutación en una muestra de ADN, una sonda ASO le sintetizarse para ser complementario a la secuencia alterada,[1] etiquetado aquí como "S". Como control, otro ASO ser sintetizado por la secuencia normal "A". Cada ASO es totalmente complementario a la secuencia de destino y se atan fuertemente, pero tiene una sola discrepancia contra su alelo no objetivo (que conduce a la interacción débil). El primer diagrama muestra cómo la sonda "S" es totalmente complementaria a la meta de "S" (superior), pero es parcialmente incompatible contra el objetivo "A" (parte inferior).
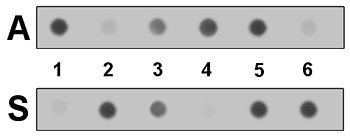
Un segmento de los genes beta-hemoglobina en la muestra QUE DNA(S) se amplificó por PCR y los productos resultantes aplicados para duplicar membranas de soporte como Dot blot. Filamentos de la DNA de la muestra se separan con álcali, y probe de cada ASO se aplica a una mancha diferente. Después de la hibridación, se utiliza un protocolo de lavado que puede discriminar entre totalmente complementarias y los híbridos no coincidentes. ASOs se lavan fuera lacras, permaneciendo los ASOs emparejados (y sus etiquetas).
En el segundo diagrama, seis muestras de ADN amplificado se han aplicado a cada uno de los dos borrones. Detección de de la etiqueta ASO que queda después de lavado permite una lectura directa de la genotipo de las muestras, cada una con dos copias del gene beta de la hemoglobina. Las muestras 1 y 4 sólo tienen el alelo "A" normal, mientras que las muestras 3 y 5 tienen alelos los "A" y "S" (y por lo tanto heterozigótica portadores de la de esto mutación recesiva). Las muestras 2 y 6 tienen sólo el alelo "S" y se verían afectadas por la enfermedad. La pequeña cantidad de 'hibridación cruzada' muestra es típica y se considera en el proceso de interpretación de los resultados finales.
Alternativas
Análisis de ASO es sólo uno de los métodos utilizados para detectar polimorfismos genéticos. Directa Secuencia de la DNA se utiliza para caracterizar inicialmente la mutación, pero es demasiado laborioso para la investigación rutinaria. Un método anterior, Polimorfismo de longitud de fragmentos de restricción (RFLP) no necesita saber la secuencia de cambio previamente, pero requiere que la mutación afecta el sitio de clivaje de un Enzima de restricción. El análisis RFLP brevemente fue adaptado para el uso de oligonucleótidos puntas de prueba,[2] pero esta técnica fue rápidamente suplantada por el análisis de la ASO de reacción en cadena de polimerasa (PCR) amplificó la DNA. La propia técnica de PCR ha sido adaptada para detectar polimorfismos, como PCR específica de alelo. Sin embargo, la simplicidad y la versatilidad del método combinado de PCR/ASO ha conducido a su uso continuado, incluso con etiquetas no radiactivo y en un formato de "reverse dot blot" donde las sondas ASO están enlazadas a la membrana y el ADN de la muestra ampliada se utiliza para hibridación.
Historia
Fue pionero en el uso de oligonucleótidos sintéticos como sondas específicas para las variaciones de la secuencia genética por R. Bruce Wallace, trabajando en la Centro médico de la ciudad de la esperanza nacional en Duarte, California. En 1979, Wallace y sus compañeros de trabajo informaron el uso de sondas ASO para detectar variaciones en un sola hebra virus bacteriano,[3] y posteriormente aplicó la técnica de clonado genes humanos. En 1983[4] y 1985[1] Laboratorio de Wallace informó la detección de la mutación para anemia de células falciformes en las muestras de la DNA genomic entera, aunque esta aplicación fue obstaculizada por la poca cantidad de etiquetas que podrían ser llevadas por la ASO.[1]
Afortunadamente también se informó de PCR, un método para amplificar grandemente un segmento específico de ADN, en 1985.[2] En menos de un año había sido emparejado PCR Análisis de ASO.[5] Esta combinación solucionó el problema del etiquetado de ASO, puesto que la cantidad de ADN diana podría ampliarse sobre un pre-existente. También, la especificidad del propio proceso PCR podría añadirse a la de las sondas ASO, lo que reduce el problema de Unión espuria de la ASO a secuencias diana no. La combinación era bastante específica que podría ser utilizado de una forma sencilla Blot dot, evitando el laborioso e ineficiente Mancha blanca /negra meridional método.
Referencias
- ^ a b c Studencki AB, BJ Conner, CC Impraim, RL Teplitz y Wallace RB «Discriminación entre los humanos beta A, beta S y genes beta C-globina usando sondas de hibridación alelo-específico del oligonucleótido.» AM J Hum Genet vol. 37, pp. 42 – 51 (1985).
- ^ a b Saiki, RK; Scharf S; Faloona F; Mullis KB; Cuerno de GT; Erlich HA; Arnheim N (20 de diciembre de 1985). "Amplificación enzimática de secuencias genómicas de beta-globina y análisis del sitio de restricción para el diagnóstico de anemia de células falciformes". Ciencia 230 (4732): 1350 – 4. doi:10.1126/Science.2999980. PMID2999980.
- ^ Wallace RB, Shaffer J, Murphy RF, Bonner J, T Hirose y Itakura K "hibridación de oligodeoxyribonucleotides sintético a Phi-X 174 DNA: el efecto de coincidencia de base par." Investigación de ácidos nucleicos, vol. 6(11), pp. 3543 – 3558 (1979).
- ^ Conner BJ, Reyes AA, Morin C, Itakura K, Teplitz RL y Wallace RB "Detección del alelo de beta globina de la S células falciformes por hibridación con oligonucleótidos sintéticos." Proc Natl Acad Sci USA. Vol. 80, pp. 278-282 (1983).
- ^ Saiki RK, Bugawan TL, cuerno GT, Mullis KB y Erlich, "Análisis de la DNA beta-globin y HLA-DQ enzimático amplificada con sondas de oligonucleótidos específicos de alelo" naturaleza vol. 324(6093) págs. 163-166 (1986).
Enlaces externos
- Imagen de autoradiogram ASO